Medidas de Dispersión
18:39 min | Última modificación: Dicimebre 13, 2021 | YouTube
[1]:
import warnings
warnings.filterwarnings("ignore")
[2]:
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
import seaborn as sns
Definición del problema
El conjunto de datos de la flor Iris contiene 150 muestras de las medidas del ancho y el largo del pétalo y del sépalo para las tres especies de esta flor (Iris setosa, Iris virginica e Iris versicolor). A partir de la muestra de datos se desean responder la siguiente pregunta:
P2.— ¿Cuál es el rango típico de la longitud y ancho del pétalo y del sépalo para cada uno de los tipos de flores?
Carga de datos
[3]:
#
# Se cargan los datos y se realiza una inspección
# inicial de la tabla y los datos
#
iris = sns.load_dataset('iris')
display(
iris.head(),
iris.tail()
)
| sepal_length | sepal_width | petal_length | petal_width | species | |
|---|---|---|---|---|---|
| 0 | 5.1 | 3.5 | 1.4 | 0.2 | setosa |
| 1 | 4.9 | 3.0 | 1.4 | 0.2 | setosa |
| 2 | 4.7 | 3.2 | 1.3 | 0.2 | setosa |
| 3 | 4.6 | 3.1 | 1.5 | 0.2 | setosa |
| 4 | 5.0 | 3.6 | 1.4 | 0.2 | setosa |
| sepal_length | sepal_width | petal_length | petal_width | species | |
|---|---|---|---|---|---|
| 145 | 6.7 | 3.0 | 5.2 | 2.3 | virginica |
| 146 | 6.3 | 2.5 | 5.0 | 1.9 | virginica |
| 147 | 6.5 | 3.0 | 5.2 | 2.0 | virginica |
| 148 | 6.2 | 3.4 | 5.4 | 2.3 | virginica |
| 149 | 5.9 | 3.0 | 5.1 | 1.8 | virginica |
P2.— ¿Cuál es el rango típico de la longitud y ancho del pétalo y del sépalo para cada uno de los tipos de flores?
Esta pregunta se refiere a una medida de la dispersión o variabilildad de los datos y a como se distribuyen.
Métricas basadas en la distancia respecto a las métricas de localización
Desviación:
Diferencia entre el valor observado y el valor estimado de localización. En modelado sus sinónimos son errores o residuales.
Rango:
Diferencia entre el valor más grande y más pequeño de la muestra de los datos.
[4]:
iris_melt = pd.melt(
iris,
id_vars="species",
var_name="Variables",
value_name="Values",
)
[5]:
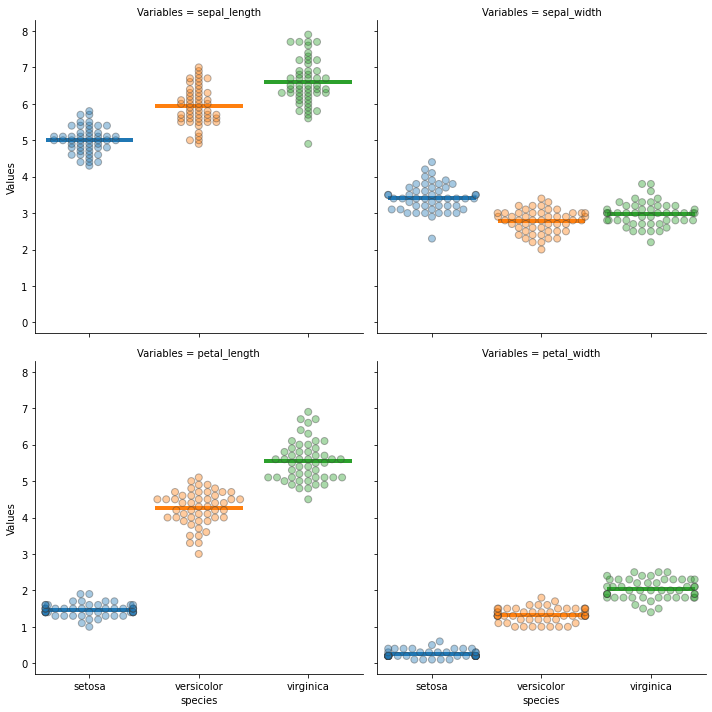
g = sns.catplot(
x="species",
y="Values",
data=iris_melt,
col="Variables",
kind="swarm",
col_wrap=2,
alpha=0.4,
s=7,
edgecolors="k",
linewidth=1,
)
colors = ['tab:blue', 'tab:orange', 'tab:green']
means = iris.groupby("species").mean()
for i_axes in range(4):
ax = g.axes[i_axes]
x_lim = ax.get_xlim()
mean_values = means[means.columns[i_axes]]
for i, (color, mean_value) in enumerate(zip(colors, mean_values)):
g.axes[i_axes].hlines(mean_value, i-0.4, i+0.4, ls='-', color=color, lw=4)
plt.show()
Varianza muestral:
[6]:
#
# Cálculo para una variable
#
from statistics import variance
display(
iris.sepal_length.var(),
variance(iris.sepal_length),
np.var(iris.sepal_length),
)
0.6856935123042507
0.6856935123042506
0.6811222222222223
[7]:
#
# Cálculo para las columnas del dataframe
#
iris.var()
[7]:
sepal_length 0.685694
sepal_width 0.189979
petal_length 3.116278
petal_width 0.581006
dtype: float64
[8]:
#
# Cálculo por especie
#
iris.groupby('species').var()
[8]:
| sepal_length | sepal_width | petal_length | petal_width | |
|---|---|---|---|---|
| species | ||||
| setosa | 0.124249 | 0.143690 | 0.030159 | 0.011106 |
| versicolor | 0.266433 | 0.098469 | 0.220816 | 0.039106 |
| virginica | 0.404343 | 0.104004 | 0.304588 | 0.075433 |
Desviación estándar muestral.
Raíz cuadrada de la varianza muestral.
[9]:
#
# Cálculo para una variable
#
from statistics import stdev
display(
iris.sepal_length.std(),
stdev(iris.sepal_length),
np.std(iris.sepal_length),
)
0.828066127977863
0.8280661279778629
0.8253012917851409
[10]:
#
# Cálculo para las columnas del dataframe
#
iris.std()
[10]:
sepal_length 0.828066
sepal_width 0.435866
petal_length 1.765298
petal_width 0.762238
dtype: float64
[11]:
#
# Cálculo por especie
#
iris.groupby('species').std()
[11]:
| sepal_length | sepal_width | petal_length | petal_width | |
|---|---|---|---|---|
| species | ||||
| setosa | 0.352490 | 0.379064 | 0.173664 | 0.105386 |
| versicolor | 0.516171 | 0.313798 | 0.469911 | 0.197753 |
| virginica | 0.635880 | 0.322497 | 0.551895 | 0.274650 |
Desviación media absoluta (MAD):
Norma L1 o distancia de Manhattan.
[12]:
#
# Cálculo para una variable
#
from scipy.stats import median_absolute_deviation
display(
iris.sepal_length.mad(),
median_absolute_deviation(iris.sepal_length)
)
0.6875555555555557
1.0378200000000002
[13]:
#
# Cálculo para las columnas del dataframe
#
iris.mad()
[13]:
sepal_length 0.687556
sepal_width 0.336782
petal_length 1.562747
petal_width 0.658133
dtype: float64
[14]:
#
# Cálculo por especie
#
iris.groupby('species').mad()
[14]:
| sepal_length | sepal_width | petal_length | petal_width | |
|---|---|---|---|---|
| species | ||||
| setosa | 0.27072 | 0.28736 | 0.13152 | 0.08256 |
| versicolor | 0.42144 | 0.25480 | 0.37920 | 0.15712 |
| virginica | 0.50256 | 0.24216 | 0.44000 | 0.22808 |
Desviación absoluta de la mediana:
Ya que la varianza y la desviación media absoluta no son robusta a outliers, se puede realizar un estimado de la desviación usando la mediana \(m\) de la muestra:
También es posible computar la varianza muestral truncada en analogía a la media truncada.
[15]:
def madm(df):
d = df._get_numeric_data().copy()
m = d.median()
for c in d.columns:
d[c] = d[c] - m[c]
return d.mad()
madm(iris)
[15]:
sepal_length 0.687556
sepal_width 0.336782
petal_length 1.562747
petal_width 0.658133
dtype: float64
[16]:
#
# Cálculo por especie
#
def madm(x):
m = x.median()
x = x - m
x = x.abs()
return x.mean()
iris.groupby('species').agg(madm)
[16]:
| sepal_length | sepal_width | petal_length | petal_width | |
|---|---|---|---|---|
| species | ||||
| setosa | 0.270 | 0.284 | 0.130 | 0.066 |
| versicolor | 0.420 | 0.250 | 0.376 | 0.154 |
| virginica | 0.492 | 0.238 | 0.440 | 0.226 |
Métricas basadas en la distribución de los datos
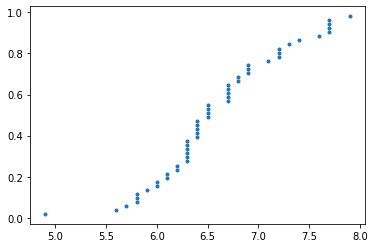
Función de Densidad Acumulada Empírica (ECDF)
[17]:
def ecdf(ax, data, param_dict=None):
if param_dict is None:
param_dict = {}
props = {
'marker': '.',
'linestyle': 'none',
}
props = {**props, **param_dict}
x = np.sort(data)
y = np.arange(1, len(data) + 1) / (len(data) + 1)
out = ax.plot(x, y, **props)
return out
_, ax = plt.subplots(1, 1)
ecdf(ax, iris.sepal_length[iris.species == 'virginica'])
plt.show()
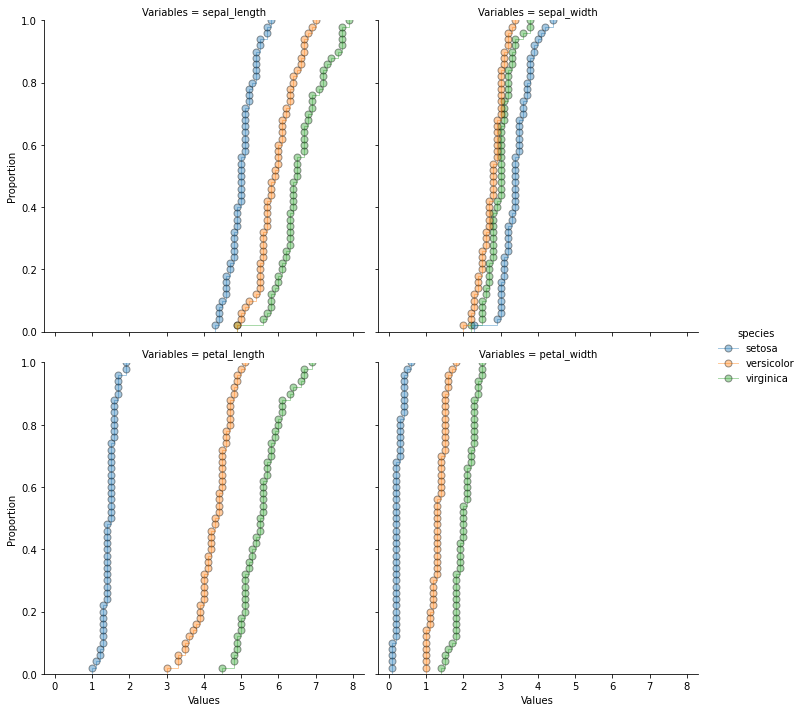
[18]:
sns.displot(
kind="ecdf",
x="Values",
hue='species',
data=iris_melt,
col="Variables",
col_wrap=2,
marker='o',
linestyle=None,
alpha=0.4,
markersize=7,
markeredgecolor="k",
linewidth=1,
)
plt.show()
Estadísticos de orden: Métricas calculadas sobre los datos ordenados.
Percentil \(p\): Valor para el cual el \(p\) por ciento de los datos son menores o iguales.
decil \(d\): Valor para el cual el \(d\) por ciento de los datos son menores o iguales. \(d\) es un múltiplo de 10.
Cuartil inferior: Valor para el cual el 25% de las observaciones son más pequeñas (percentil del 25%).
Cuartil superior: Valor para el cual el 25% de las observaciones son más grandes (percentil del 75%).
Rango intercuartil (IQR):
[19]:
#
# Cálculo de los cuartiles
#
iris.quantile(
# -------------------------------------------------------------------------
# the quantile(s) to compute
q=[0.25, 0.75],
# -------------------------------------------------------------------------
# Specifies the interpolation method to use, when the desired quantile lies
# between two data points i and j:
# {‘linear’, ‘lower’, ‘higher’, ‘midpoint’, ‘nearest’}
interpolation='linear',
)
[19]:
| sepal_length | sepal_width | petal_length | petal_width | |
|---|---|---|---|---|
| 0.25 | 5.1 | 2.8 | 1.6 | 0.3 |
| 0.75 | 6.4 | 3.3 | 5.1 | 1.8 |
[20]:
#
# Cálculo de cuartiles
#
display(
iris.quantile(0.25),
iris.quantile(0.75)
)
sepal_length 5.1
sepal_width 2.8
petal_length 1.6
petal_width 0.3
Name: 0.25, dtype: float64
sepal_length 6.4
sepal_width 3.3
petal_length 5.1
petal_width 1.8
Name: 0.75, dtype: float64
[21]:
iris.groupby("species").quantile(0.25)
[21]:
| sepal_length | sepal_width | petal_length | petal_width | |
|---|---|---|---|---|
| species | ||||
| setosa | 4.800 | 3.200 | 1.4 | 0.2 |
| versicolor | 5.600 | 2.525 | 4.0 | 1.2 |
| virginica | 6.225 | 2.800 | 5.1 | 1.8 |
[22]:
iris.groupby("species").quantile(0.75)
[22]:
| sepal_length | sepal_width | petal_length | petal_width | |
|---|---|---|---|---|
| species | ||||
| setosa | 5.2 | 3.675 | 1.575 | 0.3 |
| versicolor | 6.3 | 3.000 | 4.600 | 1.5 |
| virginica | 6.9 | 3.175 | 5.875 | 2.3 |
[23]:
iris.groupby("species").quantile(0.75) - iris.groupby("species").quantile(0.25)
[23]:
| sepal_length | sepal_width | petal_length | petal_width | |
|---|---|---|---|---|
| species | ||||
| setosa | 0.400 | 0.475 | 0.175 | 0.1 |
| versicolor | 0.700 | 0.475 | 0.600 | 0.3 |
| virginica | 0.675 | 0.375 | 0.775 | 0.5 |
[24]:
#
# El método describe de pandas computa los principales estadísticos para las
# columnas numéricas del dataframe
#
iris.describe()
[24]:
| sepal_length | sepal_width | petal_length | petal_width | |
|---|---|---|---|---|
| count | 150.000000 | 150.000000 | 150.000000 | 150.000000 |
| mean | 5.843333 | 3.057333 | 3.758000 | 1.199333 |
| std | 0.828066 | 0.435866 | 1.765298 | 0.762238 |
| min | 4.300000 | 2.000000 | 1.000000 | 0.100000 |
| 25% | 5.100000 | 2.800000 | 1.600000 | 0.300000 |
| 50% | 5.800000 | 3.000000 | 4.350000 | 1.300000 |
| 75% | 6.400000 | 3.300000 | 5.100000 | 1.800000 |
| max | 7.900000 | 4.400000 | 6.900000 | 2.500000 |
[25]:
for column in iris._get_numeric_data().columns:
print(column)
print(iris.groupby("species")[column].describe())
print("")
sepal_length
count mean std min 25% 50% 75% max
species
setosa 50.0 5.006 0.352490 4.3 4.800 5.0 5.2 5.8
versicolor 50.0 5.936 0.516171 4.9 5.600 5.9 6.3 7.0
virginica 50.0 6.588 0.635880 4.9 6.225 6.5 6.9 7.9
sepal_width
count mean std min 25% 50% 75% max
species
setosa 50.0 3.428 0.379064 2.3 3.200 3.4 3.675 4.4
versicolor 50.0 2.770 0.313798 2.0 2.525 2.8 3.000 3.4
virginica 50.0 2.974 0.322497 2.2 2.800 3.0 3.175 3.8
petal_length
count mean std min 25% 50% 75% max
species
setosa 50.0 1.462 0.173664 1.0 1.4 1.50 1.575 1.9
versicolor 50.0 4.260 0.469911 3.0 4.0 4.35 4.600 5.1
virginica 50.0 5.552 0.551895 4.5 5.1 5.55 5.875 6.9
petal_width
count mean std min 25% 50% 75% max
species
setosa 50.0 0.246 0.105386 0.1 0.2 0.2 0.3 0.6
versicolor 50.0 1.326 0.197753 1.0 1.2 1.3 1.5 1.8
virginica 50.0 2.026 0.274650 1.4 1.8 2.0 2.3 2.5
[26]:
#
# Método describe aplicado a una columna particular
#
iris.sepal_length.describe()
[26]:
count 150.000000
mean 5.843333
std 0.828066
min 4.300000
25% 5.100000
50% 5.800000
75% 6.400000
max 7.900000
Name: sepal_length, dtype: float64
[27]:
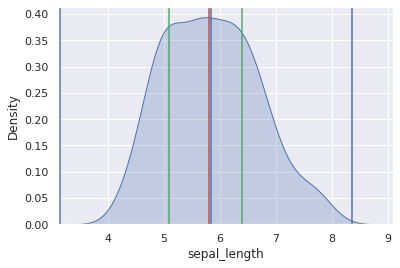
descriptive_stats = iris.sepal_length.describe()
q75 = iris.sepal_length.describe()['75%']
q25 = iris.sepal_length.describe()['25%']
iqr = q75 - q25
upper_outliers = q75 + 1.5 * iqr
lower_outliers = q25 - 1.5 * iqr
sns.set(color_codes=True)
sns.kdeplot(iris['sepal_length'], shade=True)
plt.axvline(iris.sepal_length.mean())
plt.axvline(iris.sepal_length.median(), color='r')
plt.axvline(q25, color='g')
plt.axvline(q75, color='g')
plt.axvline(upper_outliers, color = 'b')
plt.axvline(lower_outliers, color = 'b')
plt.show()
Diagramas de caja y bigote
[28]:
#
# La caja central representa los cuartiles del 25% y 75%.
# La línea centra de la caja representa la mediana
# Los bigotes están ubicados a 1.5 veces el rango interquartil.
# Los puntos por fuera de los bigotes representan outliers.
#
sns.catplot(
x="species",
y="Values",
data=iris_melt,
col="Variables",
kind="box",
col_wrap=2,
)
plt.show()
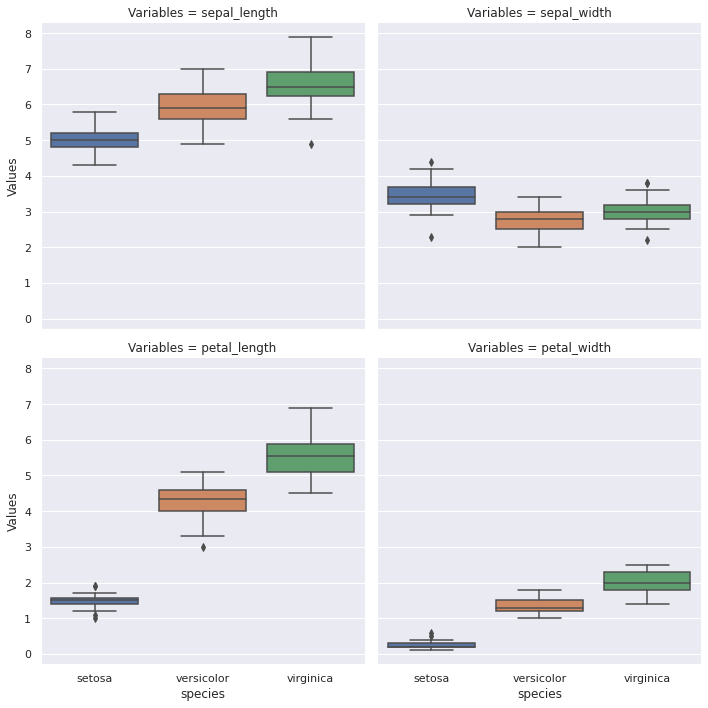
Medidas de forma
[29]:
from scipy.stats import skew
display(
iris.sepal_length.skew(),
skew(iris.sepal_length),
)
0.3149109566369728
0.3117530585022963
[30]:
from scipy.stats import kurtosis
display(
iris.sepal_length.kurt(),
kurtosis(iris.sepal_length),
)
-0.5520640413156395
-0.5735679489249765